
Machine
Learning
Joern Ploennigs
Dimensionsreduktion
Methoden zur Dimensionsreduktion werden im Machine Learning immer dann genutzt, wenn Datensätze vereinfacht werden sollen, um sie besser zu verstehen oder schneller zu erlernen. Hierbei teilt man die Verfahren in zwei Anwendungsfälle:
- Reduktion der Anzahl an Variablen in sehr großen Datensätzen mittels der Hauptkomponentenanalyse
- Reduktion der Anzahl an Beobachtungen, um typische Muster abzuleiten mittels Matrixfaktorisierung
Dimensionsreduktion wird häufig in der Vorverarbeitung von ML-Modellen eingesetzt, um etwa Overfitting zu vermeiden, die Visualisierung zu erleichtern oder Trainingszeiten zu verkürzen. So können sie zur Reduktion von Sensor-Messwerten in Brücken oder Gebäuden benutzt werden oder zur Verdichtung komplexer Materialprüfdaten auf wenige Hauptkomponenten.
Hauptkomponentenanalyse (PCA)¶
Methode¶
Die Hauptkomponentenanalyse (en. Principal Component Analysis, PCA) wird eingesetzt, um Datensätze mit vielen Variablen zu vereinfachen, um sie besser interpretieren und nutzen zu können als auch um kompaktere Modelle zu trainieren. Das ist insbesondere bei Datensätzen mit vielen, stark korrelierten Variablen sinnvoll, da sowohl Nutzer die vielen Variablen nicht verstehen können als auch die Variablen in den ML-Modellen nicht sauber getrennt werden können (zur Erinnerung: viele Modelle nehmen an das die Variablen statistisch unabhängig sind, also nicht korreliert sind).
Angenommen, wir überwachen eine Brücke mit 10 verschiedenen Sensoren. PCA hilft dabei, die wichtigsten Muster, z.B. eine Vibration in X/Y-Zugrichtung, aus diesen Daten herauszufiltern und auf 2–3 Hauptachsen zu reduzieren.
Wir betrachten den bekannten Energie- und Wetter Datensatz.
import numpy as np # Import von NumPy
import pandas as pd # Import von Pandas
egywth = pd.read_csv("../data/UROS/Energy1D_weather_clean.csv", parse_dates=[0])
egywth.head()
| Date | EV_HT_740 | EV_NT_740 | E_AV_Lab | E_SV_Lab | ES_Lab | DATUM_DT | STATIONS_ID | MESS_DATUM | QN_3 | ... | TMK | UPM | TXK | TNK | TGK | eor | TemperaturKlasse | HeizKuehlTage | QNS_4 | QNF_4 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 2020-12-30 00:00:00+00:00 | NaN | NaN | NaN | NaN | NaN | 2020-12-30 00:00:00+00:00 | 4271.0 | 20201230.0 | 10.0 | ... | 4.0 | 81.0 | 4.6 | 2.9 | 2.2 | eor | Cold | Heizgradtag | nicht alle Parameter korrigiert | 5 |
| 1 | 2020-12-31 00:00:00+00:00 | NaN | NaN | 1256.0 | 291.0 | 5.0 | 2020-12-31 00:00:00+00:00 | 4271.0 | 20201231.0 | 10.0 | ... | 3.4 | 83.0 | 4.4 | 2.3 | 0.9 | eor | Cold | Heizgradtag | nicht alle Parameter korrigiert | 5 |
| 2 | 2021-01-01 00:00:00+00:00 | 0.0 | 4080.0 | 1221.0 | 290.0 | 1.0 | 2021-01-01 00:00:00+00:00 | 4271.0 | 20210101.0 | 10.0 | ... | 2.0 | 90.0 | 3.0 | 1.1 | 0.3 | eor | Cold | Heizgradtag | nicht alle Parameter korrigiert | 5 |
| 3 | 2021-01-02 00:00:00+00:00 | 1170.0 | 2630.0 | 1243.0 | 284.0 | 2.0 | 2021-01-02 00:00:00+00:00 | 4271.0 | 20210102.0 | 10.0 | ... | 3.3 | 95.0 | 4.0 | 2.6 | 2.0 | eor | Cold | Heizgradtag | nicht alle Parameter korrigiert | 5 |
| 4 | 2021-01-03 00:00:00+00:00 | 0.0 | 3750.0 | 1222.0 | 283.0 | 2.0 | 2021-01-03 00:00:00+00:00 | 4271.0 | 20210103.0 | 10.0 | ... | 3.5 | 81.0 | 4.6 | 2.4 | 0.8 | eor | Cold | Heizgradtag | nicht alle Parameter korrigiert | 5 |
5 rows × 30 columns
Wir analysieren vom Datensatz allerdings nur die numerischen, nicht-NA Spalten, weshalb wir sie selektieren.
egywthNumber = egywth.select_dtypes(include='number').dropna()
egywthNumber = egywthNumber[['EV_HT_740', 'EV_NT_740', 'E_AV_Lab', 'E_SV_Lab', 'ES_Lab', 'FX', 'FM', 'RSK', 'RSKF', 'SDK', 'SHK_TAG', 'NM', 'VPM', 'PM', 'TMK', 'UPM', 'TXK', 'TNK', 'TGK']]
Schritt 1 - Datenzentrierung: Die Daten werden zentriert, indem der Mittelwert jeder Dimension subtrahiert wird. Dadurch sind liegen alle Daten um den Ursprung herum.
$$ \boldsymbol X_\text{cent} =\boldsymbol X − \boldsymbol μ_X. $$
egywthCentered = egywthNumber - egywthNumber.mean()
egywthCentered.head()
| EV_HT_740 | EV_NT_740 | E_AV_Lab | E_SV_Lab | ES_Lab | FX | FM | RSK | RSKF | SDK | SHK_TAG | NM | VPM | PM | TMK | UPM | TXK | TNK | TGK | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2 | -1245.840611 | 1258.984716 | 27.776201 | 16.925764 | -37.293668 | -5.778166 | -2.222162 | 1.699017 | 3.745633 | -4.973059 | -0.177948 | 2.145524 | -3.410153 | -8.547838 | -7.610699 | 12.610622 | -9.458843 | -5.770197 | -4.874891 |
| 3 | -75.840611 | -191.015284 | 49.776201 | 10.925764 | -36.293668 | -5.578166 | -3.022162 | -0.800983 | 3.745633 | -4.973059 | -0.177948 | 2.145524 | -2.510153 | -0.947838 | -6.310699 | 17.610622 | -8.458843 | -4.270197 | -3.174891 |
| 4 | -1245.840611 | 928.984716 | 28.776201 | 9.925764 | -36.293668 | 0.521834 | -0.022162 | -1.400983 | 1.745633 | -4.973059 | -0.177948 | 2.045524 | -3.410153 | 4.852162 | -6.110699 | 3.610622 | -7.858843 | -4.470197 | -4.374891 |
| 5 | 1994.159389 | -1711.015284 | 69.776201 | 21.925764 | -33.293668 | 2.521834 | 2.177838 | -1.100983 | 1.745633 | -4.973059 | -0.177948 | 2.045524 | -3.110153 | 5.752162 | -6.010699 | 6.610622 | -8.058843 | -3.870197 | -2.774891 |
| 6 | 2224.159389 | -1701.015284 | 152.776201 | 25.925764 | -37.293668 | 3.721834 | 2.377838 | 1.499017 | 3.745633 | -4.973059 | -0.177948 | 2.145524 | -2.810153 | 5.252162 | -5.910699 | 10.610622 | -7.758843 | -3.870197 | -2.574891 |
Schritt 2 - Berechnung der Kovarianzmatrix: Die Kovarianzmatrix $C$ wird berechnet, um die Streuung und Korrelation zwischen den verschiedenen Dimensionen zu erfassen.
$$ \boldsymbol C =\frac{\boldsymbol X_\text{cent}^T − \boldsymbol X_\text{cent}}{n-1} . $$
cov_matrix = np.cov(egywthCentered, rowvar=False)
Schritt 3 - Eigenwertzerlegung der Kovarianzmatrix: Die Kovarianzmatrix wird einer Eigenwertzerlegung unterzogen, um die Eigenwerte und Eigenvektoren zu berechnen. Die Eigenvektoren repräsentieren die Richtungen der größten Varianz, und die Eigenwerte repräsentieren die Größe der Varianz in diesen Richtungen. Der Eigenvektor einer quadratischen Matrix ist ein Vektor, der durch die Matrix nur um einen skalaren Faktor, den Eigenwert $λ$, gestreckt oder gestaucht wird. Mathematisch ausgedrückt:
$$ \boldsymbol C \boldsymbol v=\lambda \boldsymbol v $$
dabei ist $\lambda$ der skalare Eigenwert und $\boldsymbol v$ der zugehörige Eigenvektor.
# Berechne die Eigenwerte und Eigenvektoren
eigenvalues, eigenvectors = np.linalg.eig(cov_matrix)
Schritt 4 - Auswahl der Hauptkomponenten: Die Hauptkomponenten (Principal Components) sind die Eigenvektoren, die den größten Eigenwerten entsprechen. Diese Eigenvektoren definieren die neuen Achsen des transformierten Datenraums. Hierfür wählen wir die besten $n$ Komponenten in der Matrix $\boldsymbol W$.
# Sortiere die Eigenvektoren nach absteigenden Eigenwerten
idx = np.argsort(eigenvalues)[::-1]
eigenvalues = eigenvalues[idx]
eigenvectors = eigenvectors[:, idx]
# Wähle die n-Hauptkomponenten
n_components = 2
eigenvalues_sub = eigenvalues[:n_components]
W = eigenvectors[:, :n_components]
Schritt 5 - Transformation der Daten: Die ursprünglichen Daten werden auf die neuen Hauptkomponenten projiziert, um die transformierten Daten zu erhalten.
$$ \boldsymbol X_\text{pca} = \boldsymbol X_\text{cent} \cdot \boldsymbol W $$
# Transformiere die Daten
egywth_pca = np.dot(egywthCentered, W)
egywth_pca
array([[1764.35300097, -144.63239642],
[ -70.67495979, 177.86671771],
[1547.04272956, 99.44054371],
...,
[ 920.66526878, 888.17908265],
[ 914.15695 , 895.99134945],
[ 803.76433239, 1030.65117329]])
PCA mit SciKit-Learn¶
Natürlich gibt es dafür auch eine einfache Implementation in sklearn, die wir wie gewohnt einsetzen. Wir können zum Training fit benutzen. Wollen wir allerdings gleich die Komponenten erhalten bietet sich alternativ fit_transform an:
from sklearn.decomposition import PCA
pcam = PCA(n_components=2)
components = pcam.fit_transform(egywthNumber)
Wir können uns die Eigenwerte am Attribute singular_values_ auslesen. Es heißt singular_values_ weil die Eigenwerte nur für quadratische Matritzen definiert sind.
pcam.singular_values_
array([61197.4192215 , 11416.98508508])
Wir können uns auch mit explained_variance_ratio_ die erklärte Varianz ausgeben lassen.
print(pcam.explained_variance_ratio_)
[0.96270602 0.0335066 ]
Hier sehen wir, dass die erste Komponente mit 96.7% den Großteil der Varianz erklärt, die zweite mit 3% deutlich weniger.
Als nächstes wollen wir die Komponenten visualisieren. Das ist ein weiterer wichtiger Anwendungsbereich von PCA, da sich Datensätze mit sehr vielen Variablen schlecht visualisieren lassen, kann man sie mit PCA auf ein 2D oder 3D Dartstellung reduzieren.
import plotly.express as px
# Visualisierung der transformierten Daten
fig = px.scatter(components, x=0, y=1, width=500, height=500)
fig.update_layout( xaxis_title="Hauptkomponente 1", yaxis_title="Hauptkomponente 2")
# Visualisierung der Eigenvektoren
loadings = pcam.components_.T * np.sqrt(pcam.explained_variance_)
for i, feature in enumerate(egywthCentered.columns):
fig.add_annotation(ax=0, ay=0, axref="x", ayref="y", x=loadings[i, 0], y=loadings[i, 1], showarrow=True, arrowsize=2, arrowhead=2, xanchor="right", yanchor="top")
fig.add_annotation( x=loadings[i, 0], y=loadings[i, 1], ax=0, ay=0, xanchor="center", yanchor="bottom", text=feature, yshift=5 )
fig.show()
Was wir in dem Diagram erkennen können ist, dass es drei Cluster um "EV_NT_740", "EV_HT_740", "E_AV_Lab". Um den letzteren gruppieren sich auch die ganzen Wetterwerte, was bedeutet, dass diese deutlich stärker mit "E_AV_Lab" korreliert sind, als mit den anderen Werten.
Nichtnegative Matrixfaktorisierung (NMF)¶
Methode¶
Matrixfaktorisierung wird genutzt, um die Anzahl der Beobachtungen (die Anzahl der Variablen bleibt gleich) zu reduzieren. Dabei zerteilt man einen Datensatz in zwei niedrigdimensionale Matrizen. Das ist sinnvoll, wenn man die Daten komprimieren oder latente Merkmale extrahieren möchte. Es wird z.B. bei der Bilderkennung benutzt, aber auch als Alternative zum Clustering, wenn keine klaren Cluster erkennbar sind.
Die nichtnegative Matrixfaktorisierung ist der typischste Ansatz, wobei alle Elemente Matrizen nicht-negativ sind (sein müssen). Dies ist besonders nützlich für Anwendungen, bei denen die Daten nicht-negativ sind, wie z.B. Bilder, Texte und Empfehlungssysteme. Gegeben ist die nicht negative Matrix $\boldsymbol{X}$ mit Dimension $m × n$ welche wir in die zwei Matritzen $\boldsymbol{W}$ und $\boldsymbol{H}$ mit der Dimension $m×k$ und $k×n$ zerlegen wollen, so dass
$$ \boldsymbol{X} \approx \boldsymbol{W} \boldsymbol{H}. $$
wobei $k$ die Anzahl der latenten Merkmale ist.
Wir wollen $W$ und $H$ finden, so dass die Differenz zwischen $\boldsymbol{X}$ und $\boldsymbol{W} \boldsymbol{H}$ minimiert wird. Wir minimieren hierfür wieder den quadratischen Fehler
$$ \displaystyle \min_{\boldsymbol{W}, \boldsymbol{H}} (\boldsymbol{X} - \boldsymbol{W} \boldsymbol{H})^2 $$
Eine gebräuchliche Methode dafür ist der folgende iterative Algorithmus:
Schritt 1 - Initialisiere: Initialisiere $\boldsymbol{W}$ und $\boldsymbol{H}$ mit zufälligen nicht-negativen Werten.
Schritt 2 - Iterative Aktualisierung: Aktualisiere $\boldsymbol{W}$ und $\boldsymbol{H}$ iterativ unter Verwendung der multiplikativen Aktualisierungsregeln:
$$ \boldsymbol{H} \leftarrow \boldsymbol{H} \odot \frac{\boldsymbol{W}^T \boldsymbol{X}}{\boldsymbol{W}^T \boldsymbol{W} \boldsymbol{H}} $$
$$ \boldsymbol{W} \leftarrow \boldsymbol{W} \odot \frac{\boldsymbol{X} \boldsymbol{H}^T}{\boldsymbol{W} \boldsymbol{H} \boldsymbol{H}^T} $$
wobei $\odot$ das elementweise Produkt und $\frac{A}{B}$ die elementweise Division bezeichnet.
Schritt 3 - Konvergenzkriterium: Überprüfe die Konvergenz, z.B. ob die Änderung des Rekonstruktionsfehlers unter einen bestimmten Schwellenwert fällt oder eine maximale Anzahl von Iterationen erreicht ist.
Schritt 4 - Rekonstruktion: Die rekonstruierte Matrix $\boldsymbol{\hat X}$ ist das Produkt der Faktormatrizen $\boldsymbol{W}$ und $\boldsymbol{H}$:
$$ \boldsymbol{\hat X} = \boldsymbol{W} \boldsymbol{H}. $$
NMF in SciKit-Learn¶
Auch für die nicht-negative Matrixfaktorisierung gibt es eine Implementation in sklearn. Zu beachten ist, dass sie allerdings nur nicht-negative Werte verarbeiten kann. Deshalb müssen wir unsere Daten zuerst auf einen positiven Wertebereich normalisieren. Dafür können wir den MinMaxSkaler verwenden, der unsere Daten auf den Wertebereich $0,\ldots,1$ transformiert.
$$ \boldsymbol X_\text{mx} =\frac{\boldsymbol X − \boldsymbol \min_X}{\boldsymbol \max_X - \boldsymbol \min_X}. $$
mit dem Vektor der minimalen Werte $\boldsymbol \min_X$ und maximalen Werte $\boldsymbol \max_X$ aller Variablen in $\boldsymbol X$.
from sklearn.preprocessing import MinMaxScaler
mms = MinMaxScaler()
egywthMinMax = mms.fit_transform(egywthNumber)
mms.min_, mms.scale_
(array([ 0. , -0.14705882, -0.17349138, -0.19689119,
0. , -0.14126394, -0.08982036, 0. ,
0. , 0. , 0. , 0. ,
-0.13333333, -14.3641576 , 0.1627907 , -0.55099483,
0.09004739, 0.28315412, 0.415625 ]),
array([0.00022624, 0.00022624, 0.00053879, 0.00259067, 0.00847458,
0.03717472, 0.05988024, 0.02427184, 0.125 , 0.06198475,
0.08333333, 0.125 , 0.05128205, 0.01470156, 0.02906977,
0.01566661, 0.02369668, 0.03584229, 0.03125 ]))
Die Anwendung der NMF ist nun einfach.
from sklearn.decomposition import NMF
nmfm = NMF(n_components=2)
W = nmfm.fit_transform(egywthMinMax)
H = nmfm.components_
Hier gibt nun die Matrix $W$ aus welchen Komponenten sich jede Beobachtung in unserem Datensatz zusammensetzt. Zum Beispiel sehen wir, dass die ersten zwei Zeilen sich nur aus der Komponente 1 zusammensetzen.
pd.DataFrame(W, columns=[f'Merkmal {i+1}' for i in range(n_components)])
| Merkmal 1 | Merkmal 2 | |
|---|---|---|
| 0 | 0.237477 | 0.000000 |
| 1 | 0.241596 | 0.000000 |
| 2 | 0.223237 | 0.016886 |
| 3 | 0.222422 | 0.028341 |
| 4 | 0.252235 | 0.000574 |
| ... | ... | ... |
| 911 | 0.202307 | 0.057166 |
| 912 | 0.186173 | 0.083753 |
| 913 | 0.200195 | 0.037590 |
| 914 | 0.209245 | 0.025975 |
| 915 | 0.205023 | 0.028051 |
916 rows × 2 columns
Interessanter ist die Matrix $H$, weil sie die extrahierten Komponenten darstellen. Hierfür transformieren wir sie zurück in den originalen Datenbereich in dem wir den MinMaxScaler mit inverse_transform anwenden.
H_rescaled=mms.inverse_transform(H)
pd.DataFrame(H_rescaled, columns=egywthNumber.columns)
| EV_HT_740 | EV_NT_740 | E_AV_Lab | E_SV_Lab | ES_Lab | FX | FM | RSK | RSKF | SDK | SHK_TAG | NM | VPM | PM | TMK | UPM | TXK | TNK | TGK | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 5016.697406 | 10212.326793 | 3809.19813 | 881.823074 | 0.000000 | 45.343953 | 18.038292 | 12.541693 | 30.164496 | 0.000000 | 1.338495 | 33.113833 | 23.604693 | 1102.063064 | 36.801254 | 244.265681 | 37.890283 | 37.097455 | 50.788804 |
| 1 | 2445.902565 | 4540.833326 | 2136.22941 | 468.661514 | 199.960004 | 14.237872 | 5.890776 | 0.000000 | 0.000000 | 26.505994 | 0.000000 | 4.171526 | 25.429733 | 1063.255817 | 42.593383 | 91.811460 | 50.407605 | 36.071585 | 35.274881 |
Diese zwei Komponenten sind nun die zwei Kombinationen, welche den Datensatz am besten erklären. So lassen sich aus großen Datensätze wichtige Kombinationen ableiten. Man kann sie ähnlich wie Cluster-Zentren auffassen.
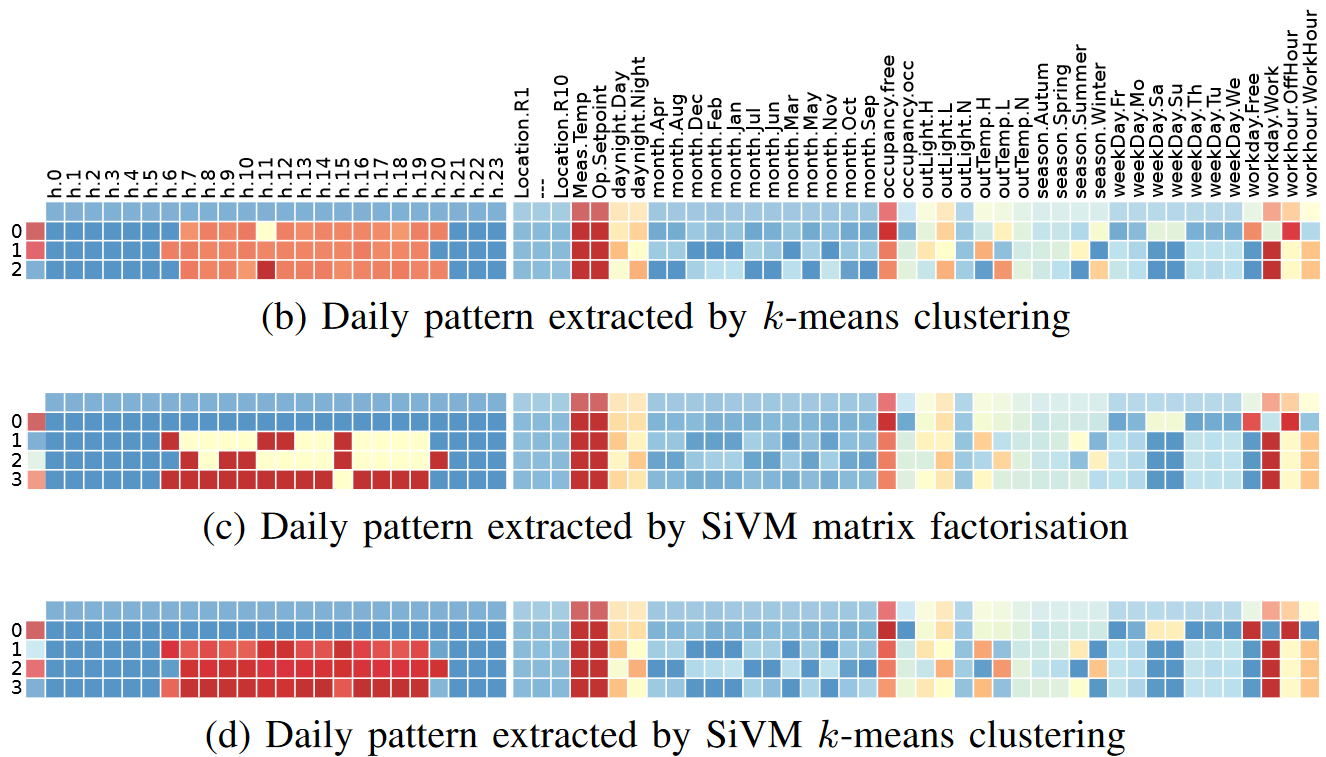
Die folgende Abbildung aus der Publikation "Understanding building operation from semantic context" {cite:p}Ploennigs15 zeigt das in einem Beispiel. In der Publikation wird die Steuerungsstragien von Gebäuden aus den Verbrauchsdaten ableitet. Die Abbildung vergleicht dabei die Muster die durch Clustering ($k$-Means), SiVM-Matrix-Faktorisierung (vergleichbar mit NMF die insbesondere charakteristische Muster identifiziert) und SiVM-Clustering (eine Kombination beider) identifiziert werden. Hierbei zeigt sich, dass die Muster, die durch SiVM-Matrix-Faktorisierung identifiziert werden deutlich ausgeprägter sind, was an der besonderen Methode SiVM liegt, zur Identifikation der Komponenten. Daraus wurden dann wieder Betriebsregeln abgeleitet durch Assoziationsanalyse, welches Entscheidungsbäumen ähneln.
und datenbanken